全基因组测序
产品描述
案例解读
全基因组测序(Whole Genome Sequencing,WGS),是对全基因组进行测序扫描,能在全基因组范围内解读所有常见和稀有变异信息,进行单核苷酸多态位点(Single Nucleotide Polymorphisms, SNP)、插入缺失位点(Insertion Deletion, InDel)、结构变异(Structure Variation,SV)以及拷贝数变异(Copy Number Variation, CNV)深度挖掘,进而全面揭示基因组突变类型与染色体重排事件,对疾病机理研究、药物靶点筛选等具有指导意义。应用范围涉及临床医药研究、群体遗传学研究、关联分析、进化分析等众多领域。
技术路线
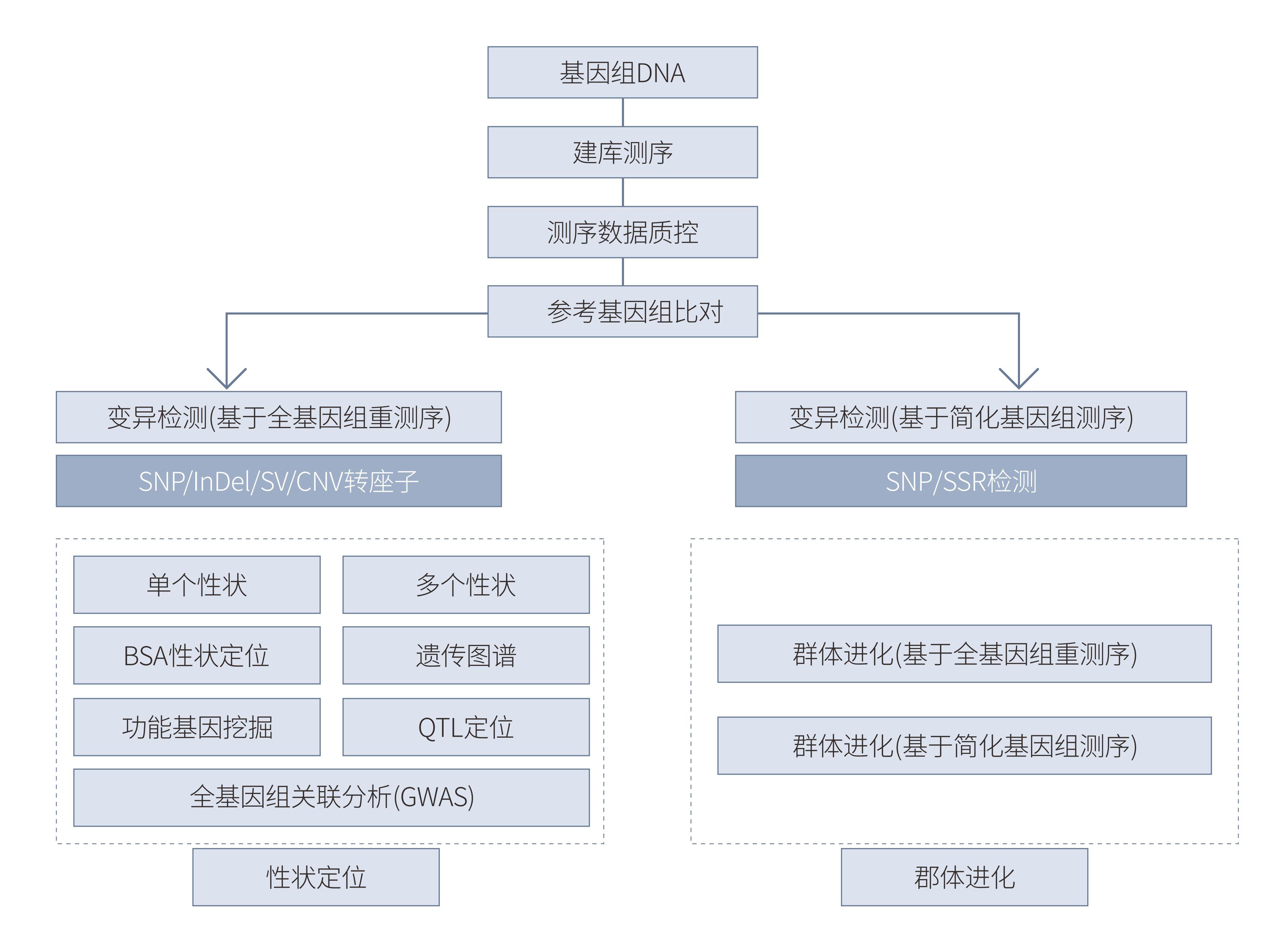
技术参数
|
样本要求 |
测序策略 |
交付周期 |
|
样品类型: 基因组DNA |
测序模式:Illumina/MGI |
45天 |
产品优势:
a.准确性高:采用Illumina /MGI平台,一次测序得到百万级别以上条数的序列信息,从几个到几十万个拷贝的精确计数;
b.检测范围广:可检测单核苷酸多态性(SNP)、插入缺失(InDel)、结构变异(SV)、拷贝数变异(CNV)等多种变异,相比芯片,可检测新的变异序列;
c.成本低耗时短:与人类全基因组从头测序相比,耗时更短、成本更低。