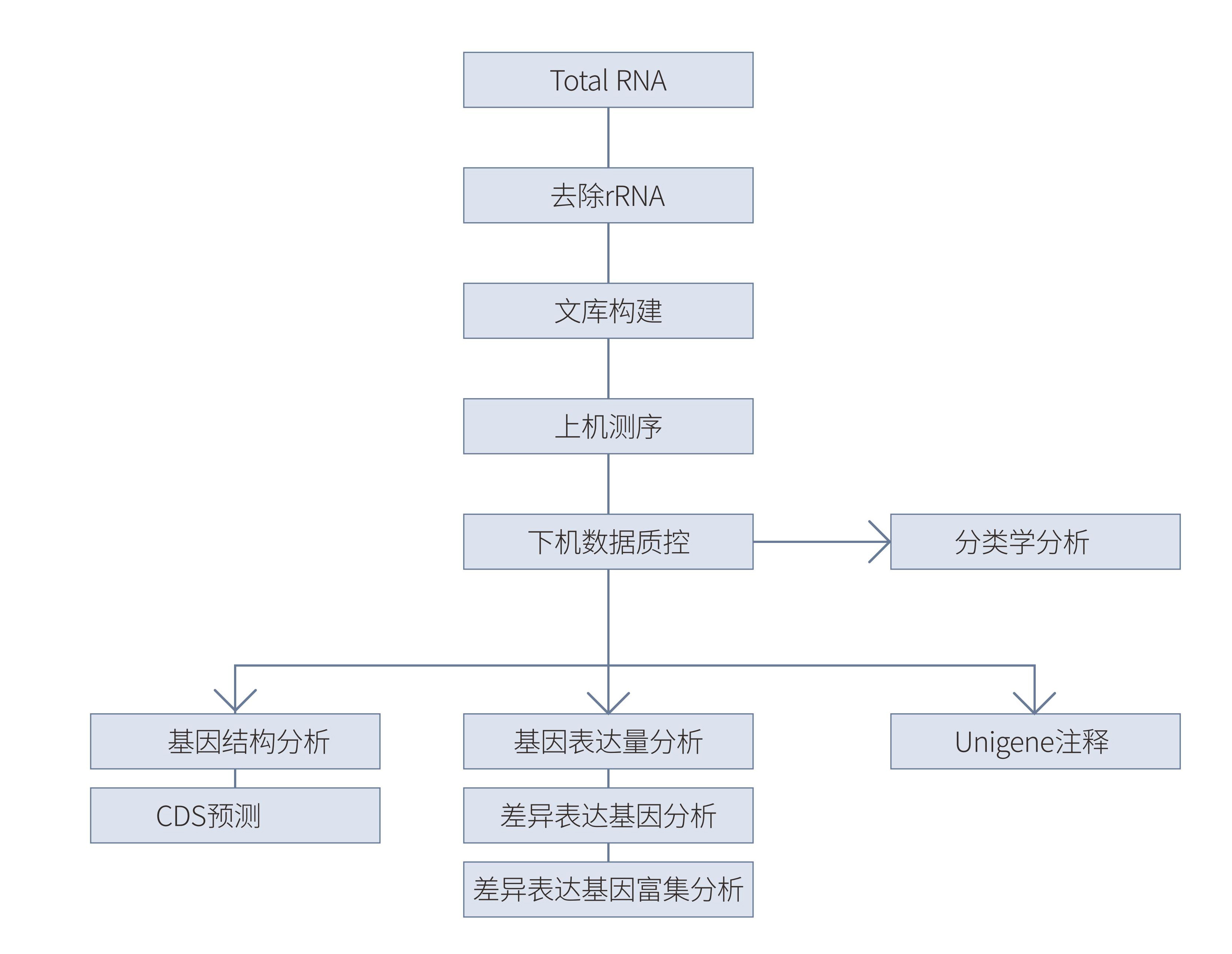
案例一 人肠道微生物宏基因组和宏转录组的整合研究
研究背景:
本研究利用受试者自己收集的人体多部位样品,结合表型分类学、宏基因组学和宏转录组(metatranscriptional)谱,进行了第一个人类微生物组的研究。结果详细描述了肠道菌群基因组潜能和基因表达之间的关系,论证了在受试者收集和运输的样品中开展宏转录组研究的可行性。
研究结果:
自采集的方法对组学研究的影响很小,可提供有代表性的宏基因组和宏转录组数据。
以KO数据库为参考,最终得到3,292个DNA和RNA相对丰度,并对DNA和RNA水平进行了关联,结果表明基因和转录本的丰度具有良好的相关性。
尽管不同个体间宏基因组的组成保守性较高,但是其在宏转录组水平却有区别,说明存在特异的宏基因组调控模式。
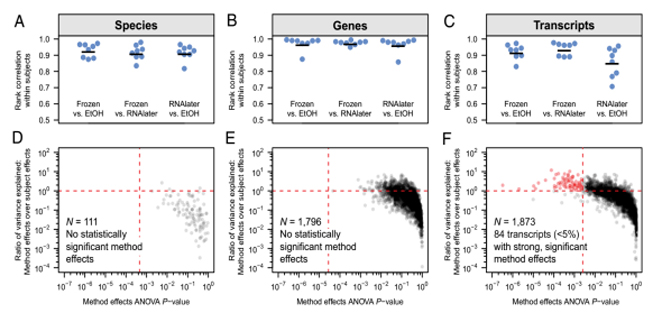
图1. 不同样本处理方法得到相同的物种组成和基因功能分类
研究结论:
本研究开发出一种新的方法,收集用于基因组和转录组分析的唾液与粪便。该方法不需要专业人员和设施,同时还能保持样品的完整性,将使我们能够不受地理限制,开展纵向研究和大规模的收集研究。这种方法也为消化道微生物组的基因组成和乳糜泻、口腔癌、牙周炎和肥胖症相关的细菌,提供了重要的见解。
案例二 宏转录组测序技术揭示草鱼肠道微生物具有分解生物质的能力
研究背景:
大多数动物只有在胃肠道中共生菌的帮助下才能分解一些特定的植物多糖。宏基因组学研究已经发现动物的胃肠道中存在着大量的分解纤维素的细菌,以及存在许多糖苷水解酶基因和碳水化合物结合基因,这些结果表明胃肠道共生菌可能参与了植物多糖的分解过程。但是,在宏基因组学研究中发现的可以分解纤维素的细菌在环境中也是大量存在的,发现的糖苷水解酶基因和碳水化合物结合基因也可能在胃肠道中没有表达。使用RNA-seq的方法可以确定这些基因是否在胃肠道中进行表达。
研究方法:
利用RNA-seq技术对11个草鱼后肠样品进行宏转录组测序,其中包括3个用鱼粮(FM)喂食的草鱼后肠样品、3个用粗纤维(CF)喂食的草鱼后肠样品、3个用苏丹草(SG)喂食的草鱼后肠样品、2个野生的草鱼后肠样品(DR、LL)。通过比较不同样品的宏转录组,揭示了草鱼后肠中微生物的物种组成、功能活性,并进一步比较了不同喂食方式下草鱼后肠宏转录组的异同。
研究结果:
1. 通过对11个样品的宏转录组测序,共获得676M 个reads(平均读长77-98 bp),62 Gb优化数据。
2.宏转录组结果表明,草鱼后肠中最主要的微生物种群属于厚壁菌门、梭菌门、变形菌门和拟杆菌门。古细菌在草鱼后肠中极少,其中最主要的古菌为产甲烷菌。不同喂食方式的草鱼后肠转录本在梭菌门和变形菌门细菌上有显著差异。
3.通过对所有转录本的直系同源蛋白簇分析,发现不同喂食方式的草鱼后肠转录本在碳水化合物转移和代谢方面有显著差异。
4.通过碳水化合物活性酶家族注释分析,发现转录本中分解胞外多糖的糖苷水解酶占了很高的比例,表明草鱼后肠微生物参与了碳水化合物的分解和代谢,从而为草鱼和微生物提供能量。
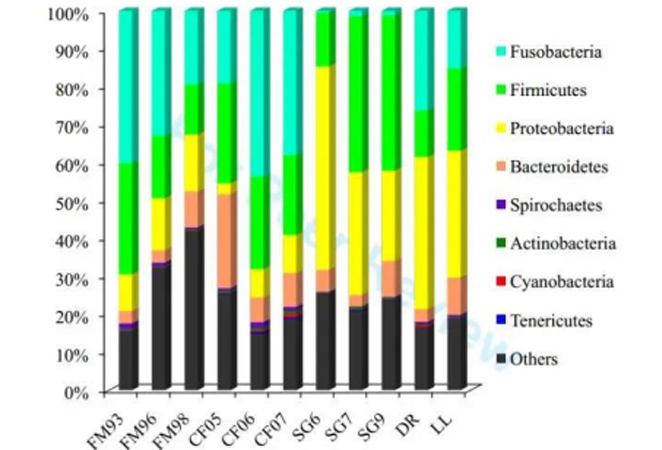
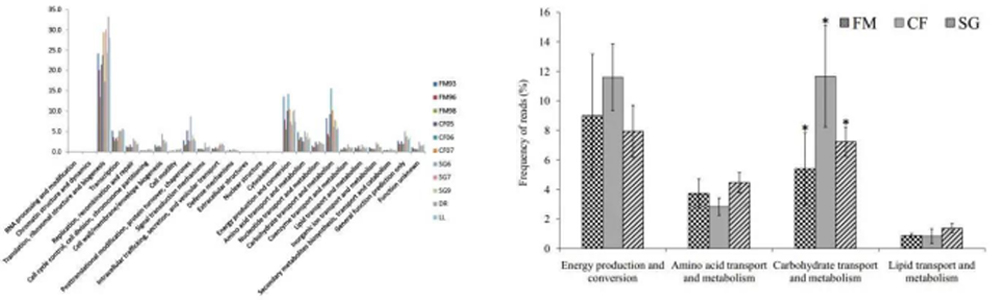
图例解读:上图,11个样品宏转录组的物种组成注释结果;左下图,11个样品宏转录组的直系同源蛋白簇(COGs)注释结果;右下图,不同喂食方式的草鱼肠道最主要的COGs丰度比较。
参考文献:
1. Franzosa E A, Morgan X C, Segata N, et al. Relating the metatranscriptome and metagenome of the human gut[J]. Proc Natl Acad Sci U S A, 2014, 111(22): E2329.
2.Shangong Wu,Yi Ren,et al.Metatranscriptomic discovery of plant biomass-degrading capacity from grasscarp intestinal microbiomes[J]. FEMS Microbiology Ecology.